# import de données
library(readr)
library(tools)
# manipulation de donénes
library(dplyr)
library(tidyr)
# travail sur les dates
library(lubridate)
# travail sur les chaines de caratcères
library(lubridate)
# visualisation
library(ggplot2)
library(ggtext)
#library(ggspatial)
# données spatiales
library(sf)
# bspline
library(splines)
# regression sparse
library(Matrix)
library(glmnet)Analyse des données IdFM
Exemple de première analyse différenciée pré / post covid
Dans ce notebook, nous allons faire quelques analyses sur les données de fréquentations journalières.
Donnees calendaires et exemple de creation de variables
Pour commencer, nous allons récupérer des informations calendaires jours fériés / vacances … et compléter notre base de données en créant quelques variables intéressantes, validations a J-7, week-end, jour de l’année, jour de la semaine,…
# lecture des données nettoyées
data.val.ok=readRDS("./data.val.ok.RDS") |>
mutate(LOG_VAL = log(NB_VALD),year=year(JOUR),yd=yday(JOUR),wd=wday(JOUR))
# statistiques globales
st_glob_stats = data.val.ok |>
group_by(ID_REFA_LDA,NAME) |>
summarise(M_VALD=mean(NB_VALD),NB_VALD = sum(NB_VALD),NB_J=n()) |>
arrange(desc(NB_VALD))
download.file("https://www.data.gouv.fr/fr/datasets/r/6637991e-c4d8-4cd6-854e-ce33c5ab49d5",destfile = "./data-raw/jf.csv")
feries = read_delim("./data-raw/jf.csv") |> filter(zone=="Métropole")
download.file("https://raw.githubusercontent.com/AntoineAugusti/vacances-scolaires/master/data.csv",destfile = "./data-raw/vacances.csv")
vacances = read_csv("./data-raw/vacances.csv") |> filter(vacances_zone_c) |> select(date,nom_vacances)
selected_ids = st_glob_stats |> ungroup() |> head(100) |> pull(ID_REFA_LDA)
#selected_ids = st_glob_stats |> filter(M_VALD>500)|> pull(ID_REFA_LDA)
N=length(selected_ids)
data.cal = data.val.ok |>
filter(ID_REFA_LDA %in% selected_ids) |>
mutate(feries=JOUR %in% feries$date,vacances=JOUR %in%vacances$date) |>
mutate(weekend=wd==6|wd==0) |>
mutate(JOUR_lag7=JOUR-7) |>
rename(id=ID_REFA_LDA)
data.cal.lag = data.cal |>
select(JOUR,id,LOG_VAL,NB_VALD) |>
rename(JOUR_lag7=JOUR,LOG_VAL_lag7=LOG_VAL,NB_VALD_lag7=NB_VALD)
data.lag = data.cal |>
left_join(data.cal.lag) |>
filter(!is.na(LOG_VAL_lag7))Exemple de modèle de régression
Pour analyser les fréquentations, nous allons construire un modèle de référence sur la période pré-pandémie. Nous allons utiliser un modèle log-linéaire du type :
model_formula=LOG_VAL~factor(wd)*id+feries+bs(yd,30)Ce modèle explique le logarithme du nombre de validation, avec un effet des jours de la semaine (wd) qui dépend de la station, un effet jours fériés commun à toutes les stations et enfin un motif annuel (yd) également commun à toutes les stations.
Nous allons tester ce modèle pour 4 stations avant de l’utiliser pour l’ensemble:
# filtrage des données
train.df = data.lag |> filter(year<=2018 , id %in% c("482368"))
fit_small = lm(LOG_VAL~factor(wd)+feries+bs(yd,24)-1,train.df)
gtsummary::tbl_regression(fit_small)| Characteristic | Beta | 95% CI1 | p-value |
|---|---|---|---|
| factor(wd) | |||
| 1 | 7.6 | 7.2, 7.9 | <0.001 |
| 2 | 8.9 | 8.6, 9.2 | <0.001 |
| 3 | 9.0 | 8.7, 9.4 | <0.001 |
| 4 | 9.1 | 8.7, 9.4 | <0.001 |
| 5 | 9.1 | 8.8, 9.5 | <0.001 |
| 6 | 9.1 | 8.7, 9.4 | <0.001 |
| 7 | 8.7 | 8.3, 9.0 | <0.001 |
| feries | |||
| FALSE | — | — | |
| TRUE | -0.83 | -0.98, -0.69 | <0.001 |
| bs(yd, 24) | |||
| bs(yd, 24)1 | 0.61 | -0.02, 1.2 | 0.058 |
| bs(yd, 24)2 | 0.62 | 0.21, 1.0 | 0.003 |
| bs(yd, 24)3 | 0.55 | 0.08, 1.0 | 0.021 |
| bs(yd, 24)4 | 0.44 | 0.03, 0.85 | 0.034 |
| bs(yd, 24)5 | 0.35 | -0.08, 0.78 | 0.11 |
| bs(yd, 24)6 | 0.45 | 0.03, 0.86 | 0.036 |
| bs(yd, 24)7 | 0.20 | -0.22, 0.63 | 0.4 |
| bs(yd, 24)8 | 0.60 | 0.18, 1.0 | 0.005 |
| bs(yd, 24)9 | 0.20 | -0.22, 0.62 | 0.4 |
| bs(yd, 24)10 | 0.46 | 0.05, 0.88 | 0.029 |
| bs(yd, 24)11 | 0.44 | 0.02, 0.86 | 0.041 |
| bs(yd, 24)12 | 0.40 | -0.02, 0.81 | 0.060 |
| bs(yd, 24)13 | 0.67 | 0.26, 1.1 | 0.002 |
| bs(yd, 24)14 | 0.51 | 0.08, 0.93 | 0.019 |
| bs(yd, 24)15 | 0.33 | -0.10, 0.76 | 0.13 |
| bs(yd, 24)16 | -0.31 | -0.73, 0.10 | 0.14 |
| bs(yd, 24)17 | 0.45 | 0.03, 0.87 | 0.037 |
| bs(yd, 24)18 | 0.40 | -0.02, 0.82 | 0.063 |
| bs(yd, 24)19 | 0.53 | 0.11, 0.96 | 0.014 |
| bs(yd, 24)20 | 0.44 | 0.02, 0.86 | 0.041 |
| bs(yd, 24)21 | 0.57 | 0.13, 1.0 | 0.011 |
| bs(yd, 24)22 | 0.17 | -0.33, 0.68 | 0.5 |
| bs(yd, 24)23 | 1.2 | 0.69, 1.7 | <0.001 |
| bs(yd, 24)24 | -0.30 | -0.80, 0.19 | 0.2 |
| 1 CI = Confidence Interval | |||
pattern_annuel=data.frame(yd=1:366,pann=predict(fit_small,data.frame(yd=1:366,wd=factor(rep(2,366,levels=1:7)),feries=rep(FALSE,366))))
ggplot(pattern_annuel)+geom_line(aes(x=as.Date("01/01/2015",format="%d/%m/%Y")+yd,y=exp(pann)))+scale_y_continuous("Composante annuelle",limits=c(0,15250))+scale_x_date("Mois",date_breaks = "months",date_labels="%m")+theme_bw()+labs(title="Composante annuelle")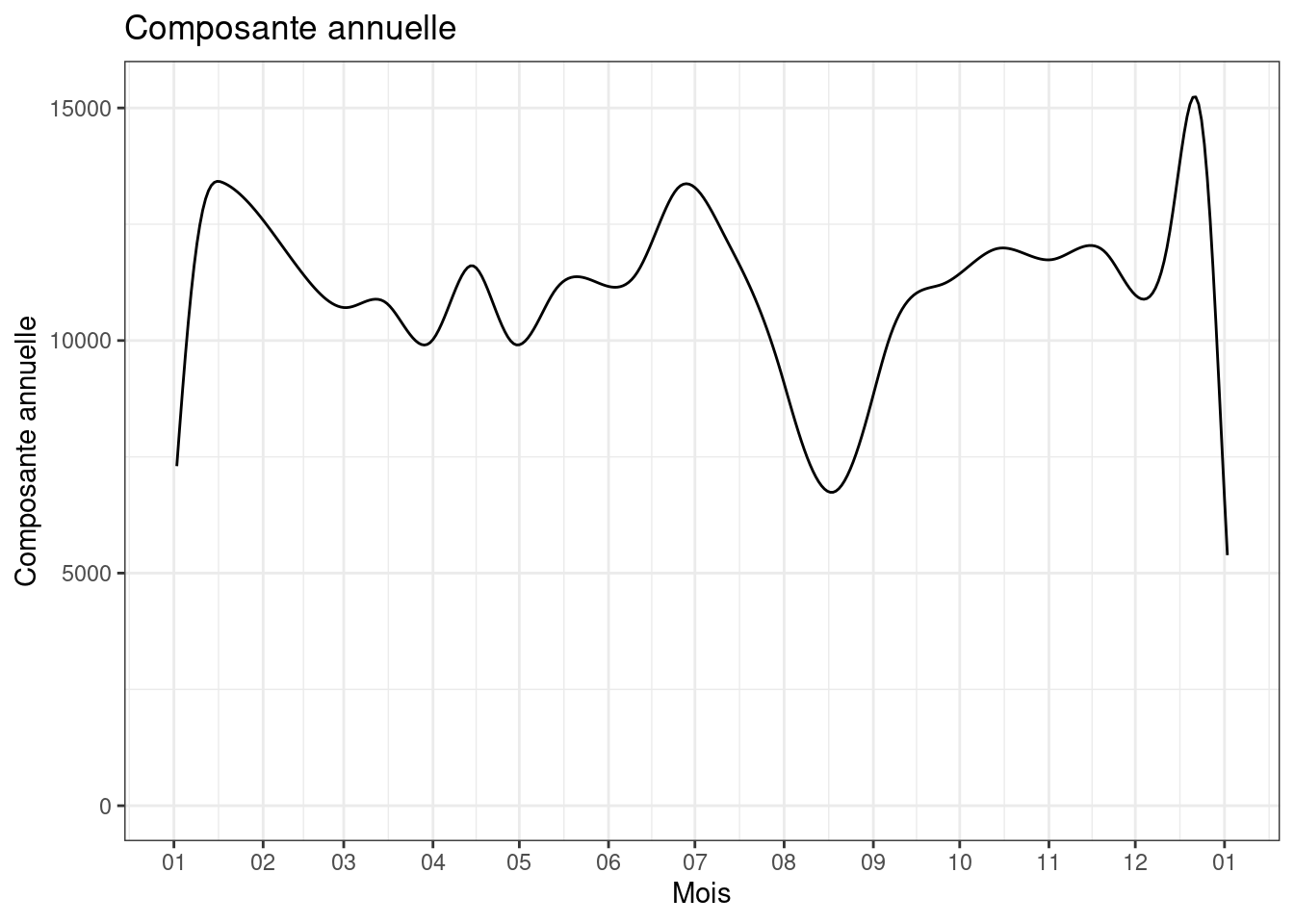
# filtrage des données
train.df = data.lag |> filter(year<=2018)
# creation de la matrice sparse
Xapp = sparse.model.matrix(model_formula,data=train.df)
# estimation
fit=glmnet(y=train.df$LOG_VAL,x=Xapp,familly="gaussian")
# prediction
yh=predict(fit,Xapp)
# residus
train.df$residuals=train.df$LOG_VAL-yh[,ncol(yh)]
train.df$NB_VAL_PRED=exp(yh[,ncol(yh)])
ids = unique(train.df$id)
subset = ids[sample(N,4,replace = FALSE)]
ggplot(train.df |> filter(id %in% subset,year==2016)) +
geom_line(aes(x=JOUR,y=NB_VALD),alpha=0.8,color="#4daf4a") +
geom_line(aes(x=JOUR,y=NB_VAL_PRED),alpha=0.8,color="#ee5555") +
facet_wrap(~paste(NAME,id))+
labs(title="Volumes <span style='color:#ee5555'>prédits</span> et <span style='color:#4daf4a'>observés</span>",
subtitle="pour une selectionde stations en 2016",y="Nombre de validations",x="Date",caption = "source: données historique de validations IdFM")+
theme_bw()+
theme(plot.title = element_markdown())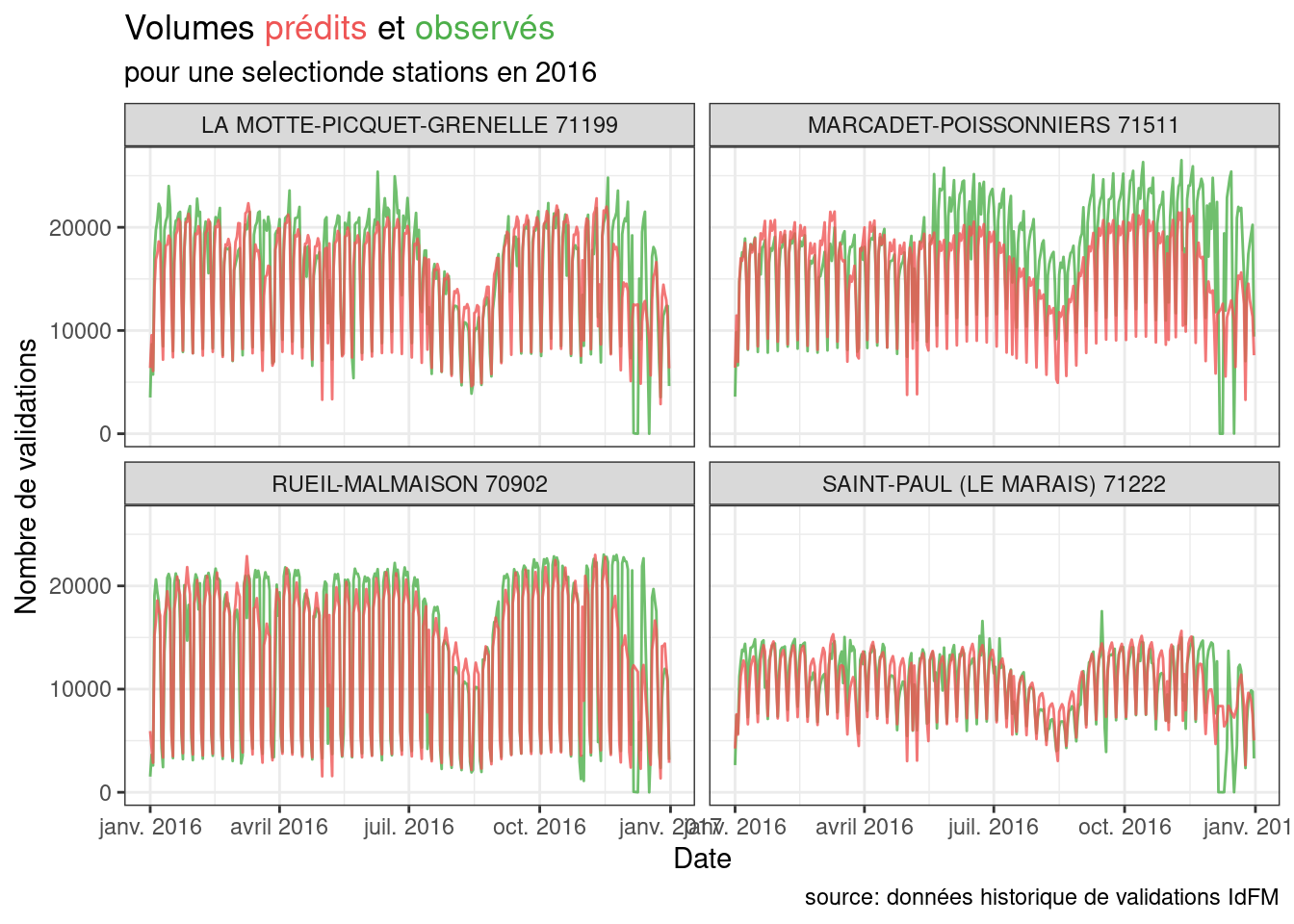
ggplot(train.df |> filter(id %in% subset,year==2017)) +
geom_line(aes(x=JOUR,y=residuals,group=paste(year,id)),alpha=0.5) +
facet_wrap(~paste(NAME,id))+
labs(title="Résidus sur les données d'apprentissage",
subtitle="pour une selectionde stations en 2017",y="log-résidus",x="Date",caption = "source: données historiques de validations IdFM")+
theme_bw()+
theme(plot.title = element_markdown())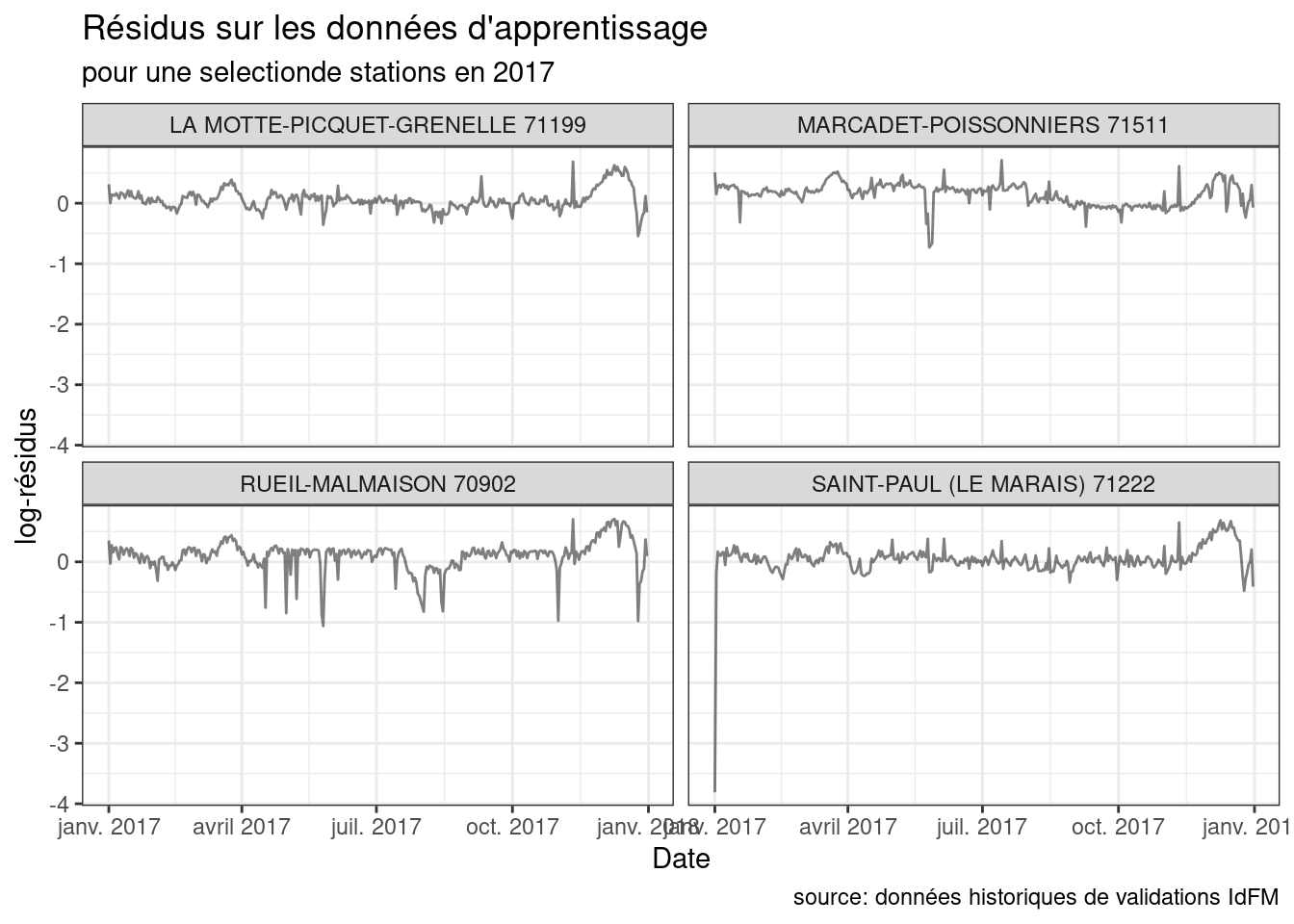
Utilisation du modèle nominale pour analyser les données post-covid
Suppression des greves / jours atypiques
Un certains nombre de jours sont très atypiques dans cette base de données d’apprentissage (grêves, …).
res.glob = train.df |>
group_by(JOUR,feries) |>
summarise(mres=mean(residuals))
ggplot(res.glob,aes(x=JOUR,y=mres))+
geom_line()+
geom_point(data=res.glob|>filter(feries),color="#4daf4a")+
geom_point(data=res.glob|>filter(abs(mres)>2),color="#ee5555")+
labs(title="Moyenne des résidus par jour",
subtitle="les <span style='color:#4daf4a'>jours ferriés</span> et <span style='color:#ee5555'>jours atypiques</span> sont mis en évidence",x="log-résidus moyen",y="Date",caption = "source: données historiques de validations IdFM")+
theme_bw()+
theme(plot.subtitle = element_markdown())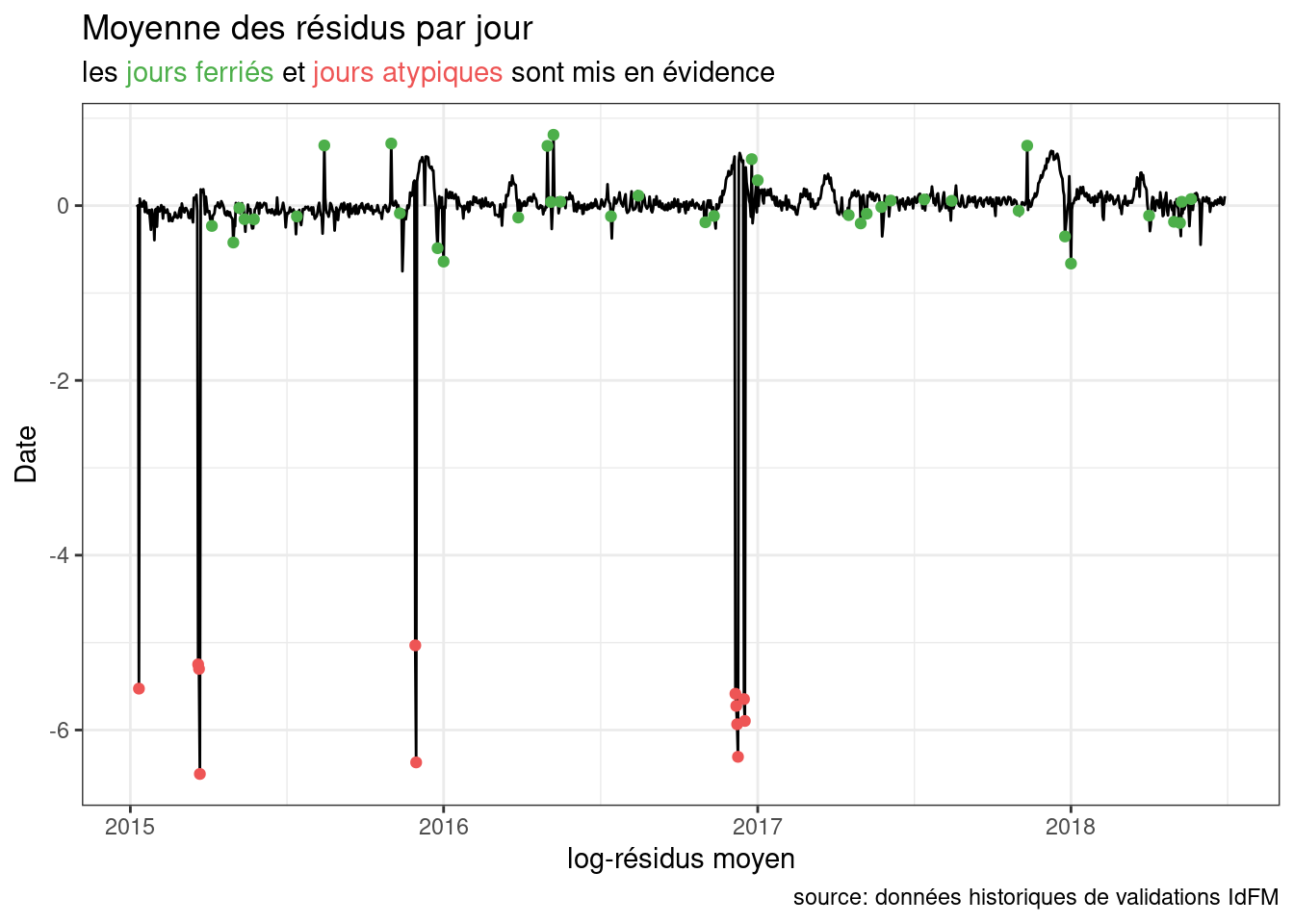
Pour ne pas que ceux-ci influencent le modèle nominale, nous allons les filtrer et ré-effectuer l’apprentissage sans ceux-ci.
greves = res.glob$JOUR[res.glob$mres<(-2)]
train.df.filtered = train.df |> filter(!JOUR %in% greves)
Xapp.filtered = sparse.model.matrix(model_formula,data=train.df.filtered)
fit=glmnet(y=train.df.filtered$LOG_VAL,x=Xapp.filtered,familly="gaussian")
yh=predict(fit,Xapp.filtered)
train.df.filtered$residuals=train.df.filtered$LOG_VAL-yh[,ncol(yh)]La régression utilisée et une régression avec pénalité L1, nous allons nous servir d’un ensemble de validation pour choisir une valeur de pénalisation.
val.df=data.lag |>
filter(year==2019, month(JOUR)<=6)
Xval = sparse.model.matrix(model_formula,data=val.df)
err=sapply(1:length(fit$lambda),\(l){
mean((val.df$LOG_VAL-predict(fit,Xval,fit$lambda[l]))^2)
})
sel.lambda = data.frame(mse=err,lambda=fit$lambda,i=1:length(err))
ggplot(sel.lambda,aes(x=log(lambda),y=mse))+
geom_line()+
theme_bw()+
labs(x="Lambda",y="Erreur quadratique moyenne",
title="Performances sur les données de validations",
subtitle="en fonction de lambda")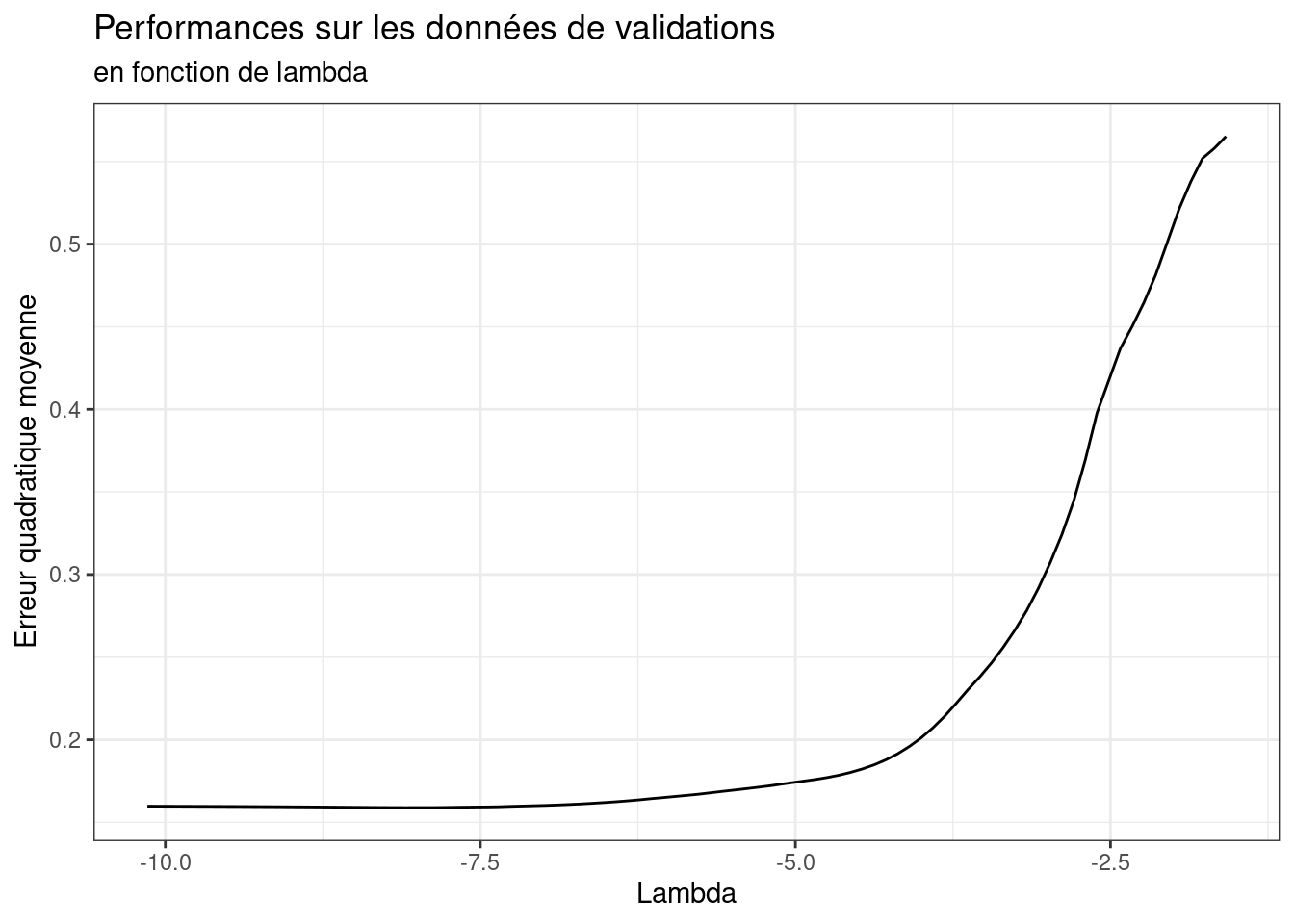
l_opt=which.min(err)Nous allons maintenant nous servir de ce denier modèle pour retirer des données post-covid les variations attendues par le modèle pré-covid.
test.df = data.lag |>
filter(JOUR>as.Date("01/07/2019",format="%d/%m/%Y"))
Xtst= sparse.model.matrix(model_formula,data=test.df)
test.df$residuals=test.df$LOG_VAL-predict(fit,Xtst,s=fit$lambda[l_opt])Cela va nous permettre d’analyser les résidus, i.e ce qui a changé depuis la pandémie:
subset = ids[sample(N,12,replace = FALSE)]
gg = test.df|> filter(id%in% subset) |> filter(year==2022)
ggplot(gg) +
geom_line(aes(x=JOUR,y=residuals,group=id),alpha=0.5)+
geom_point(data=gg |> filter(feries),aes(x=JOUR,y=residuals),color="red",size=0.3)+
facet_wrap(~paste(id,NAME))+
scale_color_discrete(guide="none")+
scale_y_continuous(limits=c(-2.5,1.5))+
theme_bw()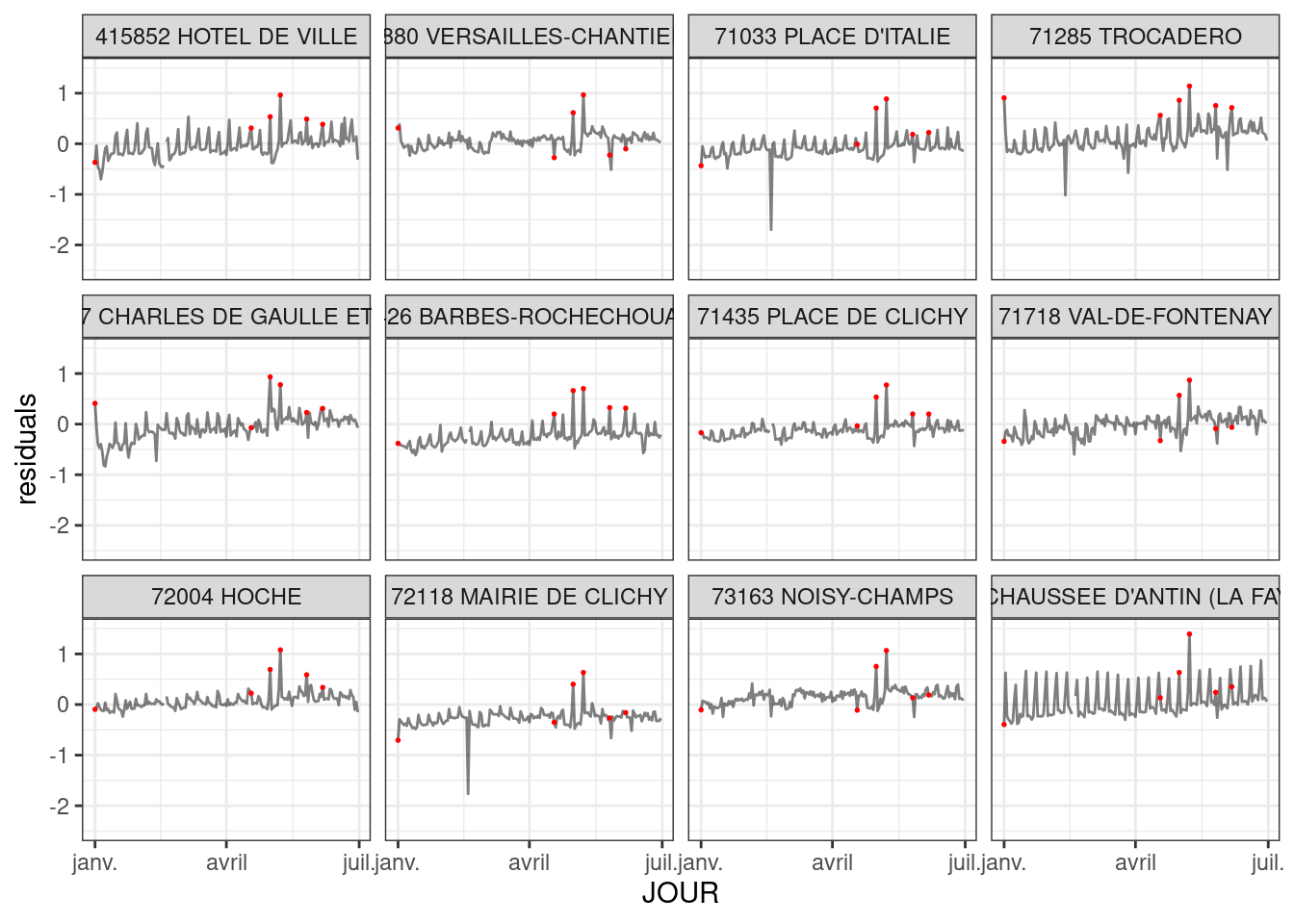
En nous focalisant sur une station et sur 2022:
selected.df = test.df|> filter(id=="482368",year>=2022)
ggplot(selected.df,aes(x=JOUR,y=residuals,group=id,label=format(JOUR,"%d/%m/%Y"))) +
geom_hline(yintercept=0,linewidth=1.5,col="#555555",alpha=0.3)+
geom_line(alpha=0.5)+
geom_point(data=selected.df |> filter(feries),color="#4daf4a")+
facet_wrap(~paste(NAME,id))+
geom_point(data=selected.df|>filter(abs(residuals)>1&!feries),col="#ee5555")+
geom_text(data=selected.df|>filter(abs(residuals)>1&!feries),vjust=-1)+
scale_color_discrete(guide="none")+
labs(title="Résidus par jour",
subtitle="les <span style='color:#4daf4a'>jours ferriés</span> et <span style='color:#ee5555'>jours atypiques</span> sont mis en évidence",y="log-résidus",y="Date",caption = "source: données historiques de validations IdFM")+
theme_bw()+
theme(plot.subtitle = element_markdown())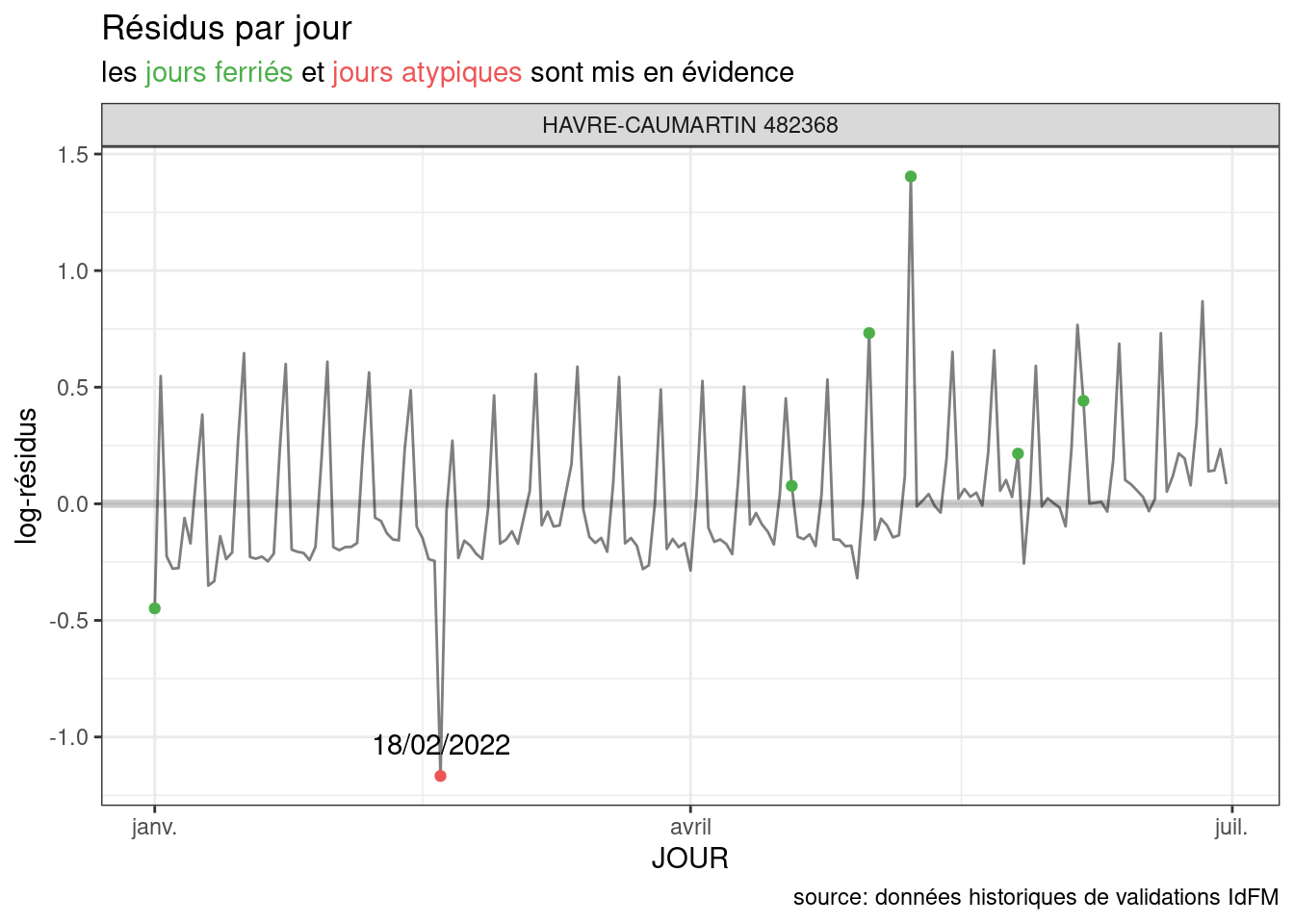
Nous pouvons souvent observer un pattern hebdomadaire dans ceux-ci. Ce que nous pouvons confirmer avec une régression.
selected.df=selected.df |>filter(!feries,abs(residuals)<1)
fit_res_wd = lm(residuals ~ factor(wd)-1,data=selected.df)
gtsummary::tbl_regression(fit_res_wd)| Characteristic | Beta | 95% CI1 | p-value |
|---|---|---|---|
| factor(wd) | |||
| 1 | 0.57 | 0.52, 0.62 | <0.001 |
| 2 | -0.10 | -0.15, -0.05 | <0.001 |
| 3 | -0.09 | -0.13, -0.04 | <0.001 |
| 4 | -0.09 | -0.14, -0.05 | <0.001 |
| 5 | -0.11 | -0.16, -0.07 | <0.001 |
| 6 | -0.16 | -0.21, -0.11 | <0.001 |
| 7 | 0.13 | 0.08, 0.17 | <0.001 |
| 1 CI = Confidence Interval | |||
pred_days=predict(fit_res_wd,data.frame(wd=factor(1:7,levels = 1:7)))
lev=c("Dimanche","Lundi","Mardi","Mercredi","Jeudi","Vendredi","Samedi")
df.wd=data.frame(evol=round(exp(pred_days)-1,3)*100,wd=factor(lev,levels=c(lev[2:7],lev[1])))
ggplot(df.wd,aes(y=wd,x=evol,fill=evol))+geom_col()+
theme_bw()+
scale_fill_distiller(palette="RdBu",breaks=c(-15,-5,0,5,50),values=scales::rescale(c(-15,-5,0,5,50)))+
labs(x="Evolution (%) par rapport à la période prè-COVID",y="",title="Evolution de la fréquentation suivant le type de jour")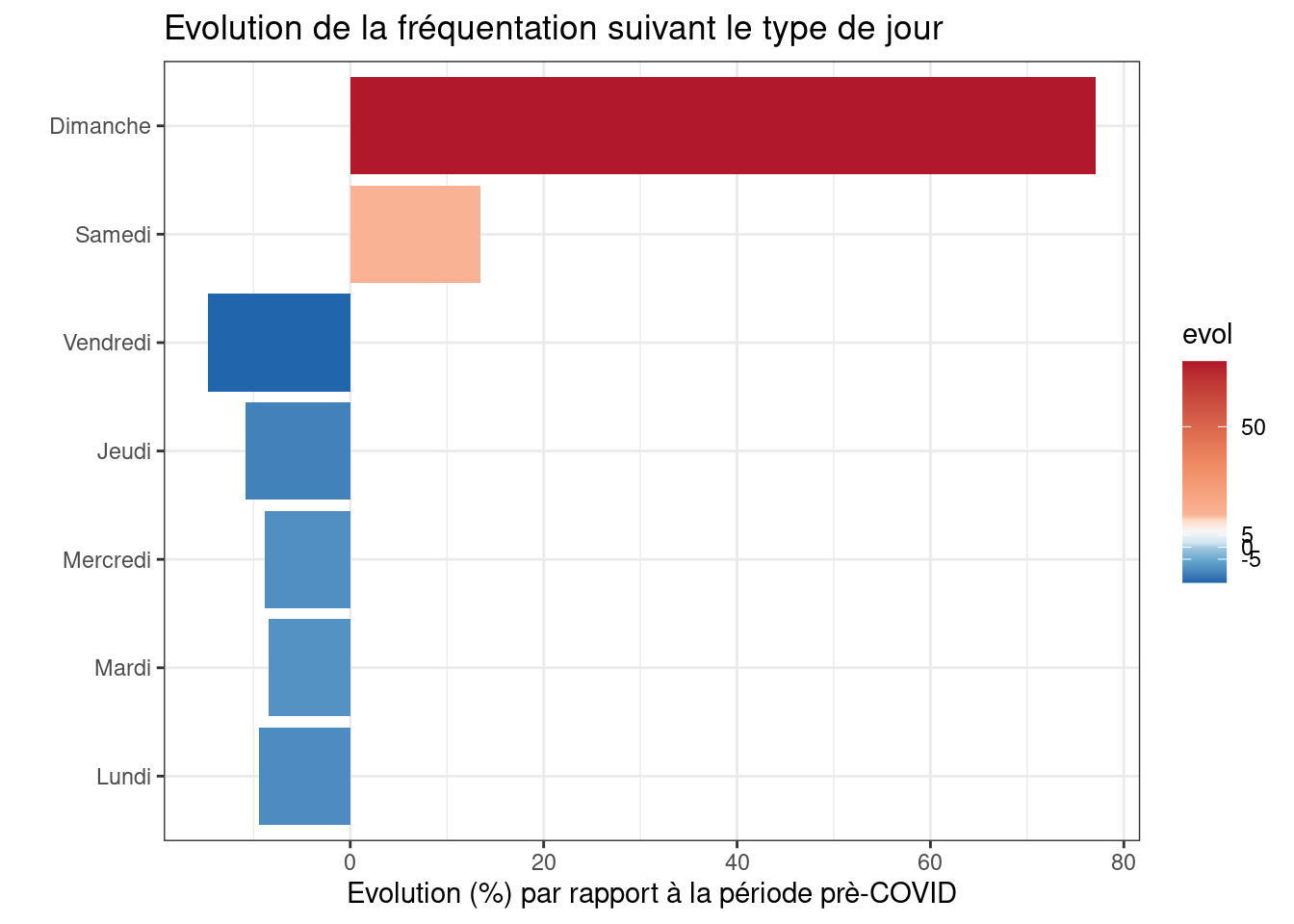
conf_intervals=round((exp(confint(fit_res_wd,level=0.95))-1)*100,1)
df.wd=cbind(df.wd,conf_intervals)
ggplot(df.wd)+geom_errorbar(aes(x=wd,ymin=`2.5 %`,ymax=`97.5 %`))+
geom_hline(yintercept=0,color="red")+
geom_point(aes(x=wd,y=evol))+
labs(y="Evolution en % par rapport au modèle pré-covid",x="Jour de la semaine",title="Evolution en % par rapport au modèle pré-covid")+
theme_bw()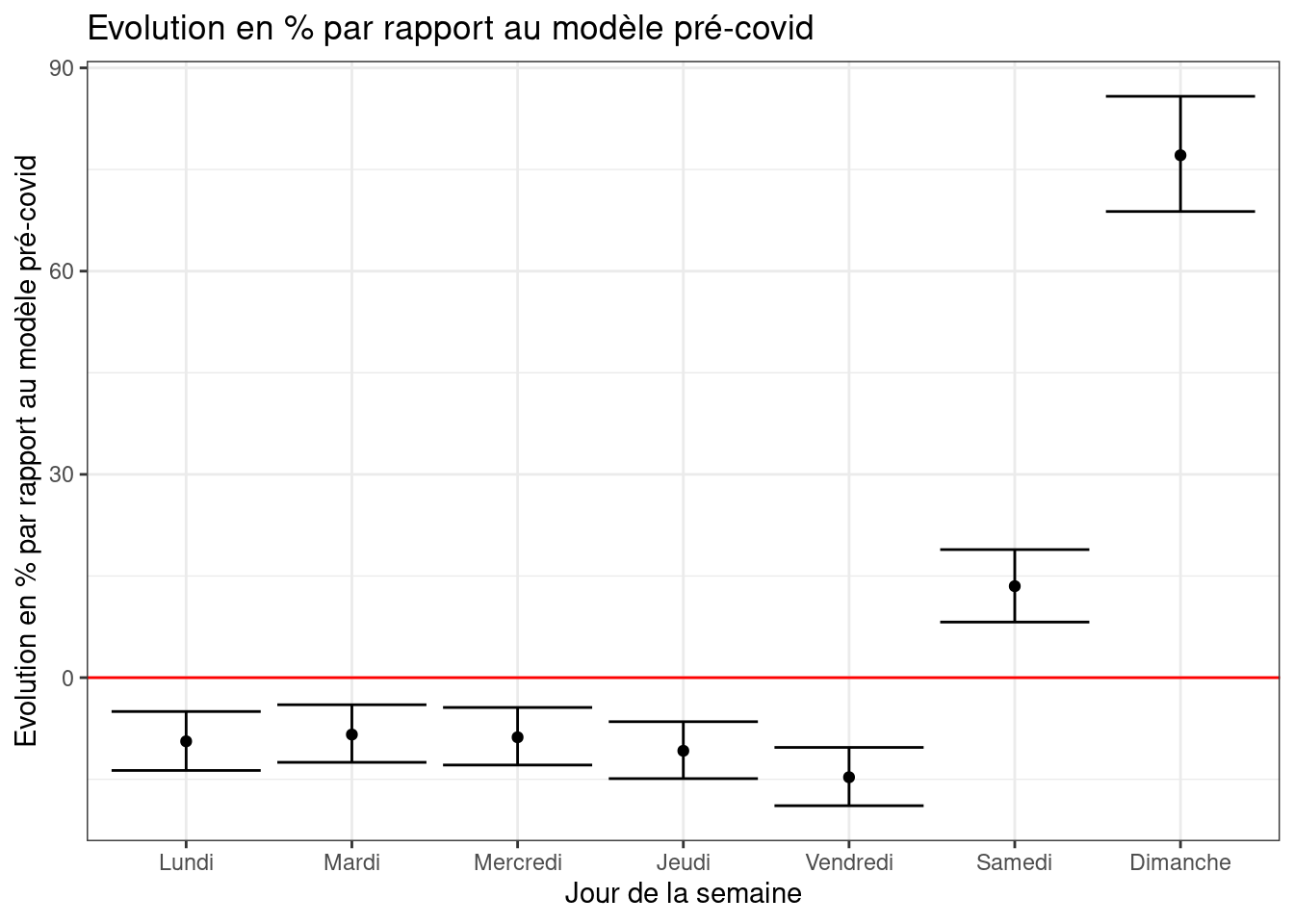
gg.cal=selected.df |> mutate(y=forcats::fct_rev(wday(JOUR,label=TRUE,week_start = 1))) |>
mutate(x=week(JOUR),md=mday(JOUR),month=month(JOUR,label=TRUE)) |>
select(x,y,md,month,year,residuals)
col_scale_vals = c(seq(min(gg.cal$residuals),0,length.out=5)[1:4],0,seq(0,max(gg.cal$residuals),length.out=5)[2:5])
month = gg.cal |> filter(md==1) |> filter(!duplicated(x)&!duplicated(month))
ggplot(gg.cal,aes(x=x,y=y,fill=residuals,label=md))+geom_tile(color="#000000")+
coord_equal(expand = FALSE)+
scale_fill_distiller("log-residuals",palette="RdBu",values=scales::rescale(col_scale_vals))+
geom_text(size=2,hjust=-0.25,vjust=-0.25)+
scale_x_continuous("",breaks=month$x,labels = month$month)+
theme_bw()+
facet_grid(year~.)+
labs(y="",title="Calendrier des résidus")+
theme(legend.position="bottom")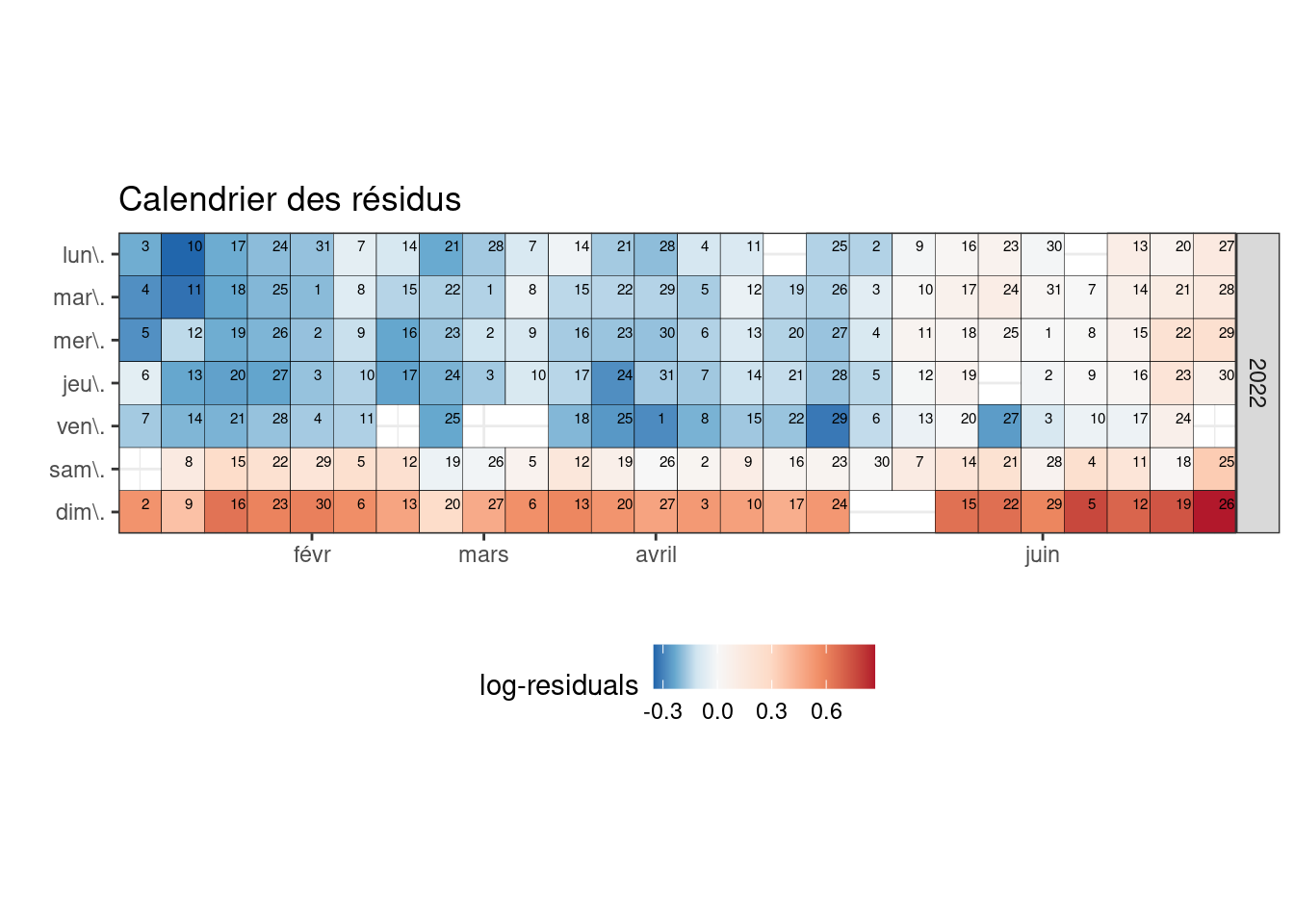
Application shiny
Pour explorer plus facilement ces résultats une petite application shiny est disponible, à vous de jouer !